
Ángel A. Islas Descargar versión PDF
Eduardo Salinas Stefanon
“Me inclino a creer que la vida tal como se nos presenta debe ser una función de la asimetría del universo o de las consecuencias que ésta implica”. Louis Pasteur. [1]
La segunda ley de la termodinámica dicta que todo sistema en el universo tiende al equilibrio en el cual la temperatura, la presión y el potencial químico tienden a ser uniformes; sin embargo, para toda célula que conforma un tejido, es vital mantener una concentración desigual de substancias en su interior y en el exterior. Específicamente, la desigualdad de cargas eléctricas a través de la membrana de una célula permite a esta establecer un no equilibrio dinámico que oscila alrededor de un estado estable desigual el cual, mediante una sofisticada maquinaria molecular delicadamente acoplada, es capaz de reaccionar apropiadamente a una serie de variaciones azarosas para regresar a tal estado estable.
Las bases experimentales y teóricas de dicha oscilación eléctrica en las células excitables fueron descritas por primera vez por Hodgkin y Huxley en 1952; más tarde, el desarrollo del entendimiento de procesos termodinámicos irreversibles y las valiosas aportaciones de Turing (sobre patrones espaciales en sistemas químicos), Lotka y Volterra (sobre oscilaciones ecológicas predador-presa) y Balescu (sobre cinéticas no lineares en oscilaciones de sistemas químicos abiertos), permitieron a Ilya Prigogine, premio Nobel de química en 1977, describir la autoorganización temporal que este tipo de comportamientos originan. [2 ]
En términos generales, un sistema abierto –como la célula– que intercambia materia y energía con su entorno, frecuentemente opera lejos del equilibrio y eventualmente alcanzará un nuevo estado estable. A consecuencia de procesos de retroalimentación su cinética es de naturaleza no-linear y eventualmente generará propiedades emergentes de autoorganización constituyendo lo que Prigogine llama una estructura disipativa. [2]
En el caso de las células excitables, al estado estable o de reposo lo caracteriza una asimetría en la distribución de cargas, ya que en el interior de la célula predomina una carga eléctrica negativa con respecto al exterior celular, lo que se conoce como potencial de membrana o de reposo. Las variaciones en este potencial eléctrico por medio del intercambio de partículas inorgánicas con carga constituyen la manera a través de la cual las células despliegan su capacidad de autoorganización temporal. Por medio de este comportamiento dinámico regenerativo, lejano al equilibrio, la célula censa el ambiente, recibe y transmite información codificada. Este fenómeno es de fundamental importancia ya que el funcionamiento de todo ser vivo depende de la capacidad con la que las células que lo componen se comuniquen.
La secuencia de eventos moleculares por los cuales el potencial de membrana se incrementa rápidamente (despolarización), y disminuye nuevamente (repolarización), hasta alcanzar su estado estable inicial, sigue una trayectoria estereotípica y se conoce como potencial de acción. El desencadenamiento y el acople temporal de este fenómeno dependen de proteínas altamente especializadas y selectivas las cuales permiten el paso de iones positivos y negativos a través de la membrana de la célula en respuesta a cambios físicos y químicos. Estas macromoléculas son llamadas canales iónicos.
LOS CANALES IÓNICOS: MÁQUINAS DE LENGUAJE CELULAR
A diferencia de otras proteínas acarreadoras de moléculas como las bombas y transportadores secundarios (e.g. de nutrientes), que precisan de un gasto de energía metabólica (transporte activo), los canales iónicos se valen del gradiente electroquímico para transportar iones; es decir, la fuerza impulsora que dirige el flujo iónico tiende a igualar las concentraciones y cargas en ambos compartimentos, interior y exterior celular, lo que se denomina transporte pasivo; este es mucho más rápido, pues miles de iones son transportados en milésimas de segundos; importantemente, no precisa de un gasto extra de energía: en principio, la segunda ley de la termodinámica le basta.
La célula, a lo largo de la evolución de la vida, ha generado mecanismos de retroalimentación (e.g. repolarización) situando al sistema en condiciones de no equilibrio, el cual, como indica Prigogine, crea “correlaciones de largo alcance” [2] por medio de las cuales la célula coordina su funcionamiento interno y su comunicación con otras células en un tejido.
Para tal efecto entra primero en juego un factor fundamental; la composición y estructura de los canales le confieren dos facultades primordiales: selectividad y la regulación de su actividad. La selectividad refiere al hecho de que los canales son más permeables a un solo tipo de ión, siendo los más importantes el potasio, el sodio, el calcio (con cargas positivas) y el cloro (con carga negativa). Los canales pueden existir en un estado abierto o cerrado y las transiciones entre estos estados se encuentran reguladas ya sea por ligandos, es decir, otras moléculas que al unirse a ellos abren o cierran el canal, o por voltaje, cuando los canales abren o cierran en respuesta a diferentes potenciales eléctricos a través de la membrana en la que se encuentran insertos.
La primera fase ascendente del mecanismo por el cual las células establecen comunicación es llamada despolarización. En ella los canales de sodio dependientes de voltaje desempeñan un papel esencial, siendo los responsables de la generación y propagación de potenciales de acción en neuronas, células musculares y neuroendócrinas [3] (Figura 1).
El estudio de su estructura y función es de gran importancia biológica y médica, ya que la expresión y/o actividad anormal de estos canales han sido identificadas como un factor determinante en un sinnúmero de enfermedades llamadas canalopatías, que incluyen arritmias cardiacas, epilepsia, dolor neuropático e inflamatorio, parálisis muscular, entre otros.[4]
Se estima que un 13.4% de los medicamentos actuales ejercen su acción terapéutica sobre un canal iónico, haciéndolos el segundo mayor blanco terapéutico molecular (después de los receptores acoplados a proteínas G).[5]
EXPERIMENTOS VIRTUALES
El diseño de fármacos y el estudio de la relación entre su estructura y sus propiedades biológicas data del siglo XIX, destacando la aportación de John Langley, Paul Ehrlich y Alfred Clark, responsables del desarrollo del concepto de receptor, el cual se define como la macromolécula blanco sobre la cual el fármaco (o droga) ejerce su acción.
Sin embargo no fue hasta los años 50’s del siglo XX, que Hansch propone aplicar cálculos y estadística para establecer relaciones cuantitativas entre la estructura y la actividad de moléculas biológicamente activas. Más tarde, con el advenimiento de nuevas tecnologías como las gráficas por computadora en los 80’s y 90’s surge el modelaje molecular.[6]
El modelaje molecular consiste en un conjunto de métodos computacionales que además de ser empleados para describir y analizar sistemas y fenómenos naturales a escala atómica, son aplicados en el campo de la farmacología, en el descubrimiento y optimización de nuevas moléculas (o ligandos) con afinidad a un blanco biológico (o receptor) sobre el cual ejerzan un efecto terapéutico. Dentro de estos métodos, el cribado virtual (detección de ligandos a partir del receptor), depende de la disponibilidad de información estructural del blanco biológico, la cual puede ser determinada experimentalmente o derivada computacionalmente.[7]
Este tipo de experimentación virtual, es decir, llevada a cabo por computadora, se denomina in silico. Aunque el desarrollo de estos experimentos teóricos es mecanicista y está arraigado intrínsecamente a una visión reduccionista y determinista de los sistemas vivos, existen diversas ventajas frente a métodos tradicionales, por ejemplo, la reducción en el número de moléculas a ser sintetizadas o probadas, la agilización de la experimentación a través de la predicción de la mayoría de propiedades fisicoquímicas (Figura 2) y farmacéuticas de cualquier molécula con tan solo contar con su fórmula, y la capacidad masiva de procesamiento de información, lo que se traduce en la reducción de la experimentación en animales y de consumo de recursos de laboratorio en general.
LA ESTRUCTURA DETERMINA LA FUNCIÓN
Como se ha mencionado anteriormente, los canales iónicos constituyen blancos terapéuticos importantes, componen el 1.5% del genoma humano y se calcula que las ventas mundiales de medicamentos que actúan sobre estas proteínas están sobre los 12 billones de dólares.[5]
No obstante, la mayoría de estos medicamentos han sido descubiertos mediante métodos tradicionales empleando animales o tejidos sin conocimiento previo de su blanco molecular.[5]
A la luz del desarrollo tecnológico y del avance de la biología molecular y el entendimiento de procesos fisiopatológicos, es imprescindible conocer a detalle, en primera instancia, la actividad biológica y el funcionamiento normal de los canales iónicos, y en segundo, su farmacología, es decir, cómo modifican su actividad los medicamentos y drogas tanto en procesos patológicos como en organismos sanos. Sin embargo, para todo lo anterior es necesario disponer de información confiable acerca de su estructura y su actividad.
Como la mayoría de canales iónicos, los canales de sodio dependientes de voltaje son complejos proteicos formados por una subunidad (α) que al atravesar la membrana de la célula forma un poro, y las subunidades (ß) accesorias que modifican la actividad del canal. Importantemente, su secuencia (i.e. las biomoléculas llamadas aminoácidos que la componen), varía según el tipo de célula en la que se encuentren.
Así, por ejemplo, la actividad moduladora que ejerce la subunidad ß1 en el canal es significativa en el sistema nervioso y en el músculo esquelético, pero no en el corazón.[3]
Se conoce que la mutación de uno solo de los cerca de 2000 aminoácidos que componen la subunidad α es causa de enfermedades tales como la miotonía, en la que una mutación puntual (el cambio de un aminoácido por otro en la secuencia, i.e. V445M) del canal de sodio de músculo esquelético produce un desorden muscular en el cual al paciente se le dificulta relajar sus músculos después de flexionarlos.[8] Igualmente, en la insensibilidad al dolor congénita (CIP) se conocen varias mutaciones “sin sentido” (e.g. R277X, Y328X, W897X) sobre la secuencia del canal de sodio del sistema nervioso periférico, que producen en el individuo la incapacidad de percibir los estímulos dolorosos.[9]
En ambas patologías, un cambio estructural ha alterado la actividad del canal de sodio: se ha modificado uno o más parámetros en la cinética de conducción de cargas positivas (iones sodio) al interior celular, lo cual altera el potencial de acción y, por ende, la comunicación entre células.
En el primer caso, la mutación V445M incrementa la corriente de sodio, el canal permanece más tiempo activo o abierto y se prolonga la despolarización en el potencial de acción y, por tanto, se retrasa la relajación del músculo después de una contracción.
En el segundo caso las mutaciones “sin sentido” producen el truncamiento de la proteína que forma el canal, volviéndolo no funcional. Al registrarse un estímulo doloroso, la información sobre este estímulo precisa de codificarse y transmitirse en forma de potenciales de acción a través del sistema nervioso periférico al cerebro, pero ya que el canal de sodio no es funcional, los potenciales de acción no pueden iniciarse y la información sobre el estímulo doloroso nunca llega al cerebro.
ONTOGENIA DE UN MODELO
Pero no todas las mutaciones puntuales modifican la actividad del canal tan significativamente, por lo que se pretende identificar a los aminoácidos que participan principalmente en los eventos moleculares que subyacen a los procesos fisiológicos.
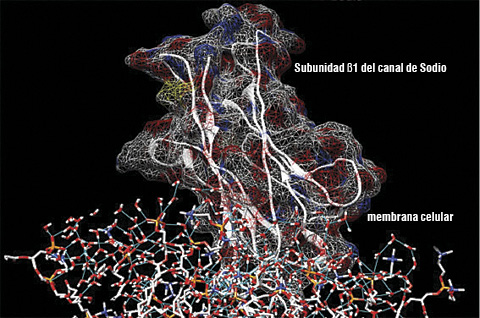
En el Laboratorio de Biofísica Cardiaca del Instituto de Fisiología de la BUAP pretendemos identificar a los aminoácidos involucrados en la asociación molecular que determina la modulación que ejerce la subunidad ß1 del canal de sodio (Figura 3).
Las proteínas α y ß1 provienen de dos genes diferentes que a lo largo de la evolución han establecido una asociación funcional.
Para tal efecto se ha desarrollado un modelo 3D a escala atómica de la subunidad ß1, por medio de un método in silico conocido como modelaje molecular por homología.
Esto consiste en: a) identificar una proteína estrechamente relacionada por evolución divergente de una proteína ancestral común con la proteína blanco a modelar, la cual haya sido determinada experimentalmente (molde). b) alinear ambas secuencias para c) mutar computacionalmente cada aminoácido de la proteína blanco en el lugar correspondiente de la estructura resuelta experimentalmente.
Por último, el modelo se refina y evalúa de acuerdo a las leyes fisicoquímicas que determinan la geometría molecular. Una vez que se cuenta con información estructural confiable sobre el canal, con principios fisicoquímicos y resultados experimentales publicados, se establecen hipótesis sobre las posibles interacciones moleculares y cambios conformacionales que se llevan a cabo durante el mecanismo de compuerta del canal.
Aunque existen otros métodos indirectos para medir la actividad del canal (como los ensayos de unión, de flujo iónico, las sondas fluorescentes, entre otros), el estándar de oro para estudiar la actividad y farmacología de un canal son los registros electrofisiológicos, [10] los cuales miden en tiempo real la actividad del canal en función de la corriente eléctrica macroscópica (producto de la actividad de una población de canales) que se genera al variar experimentalmente el voltaje a través del tiempo.
Mediante diferentes protocolos de estimulación y en diversos tipos de preparaciones se obtienen parámetros estadísticos que describen la cinética del canal, sus tiempos y voltajes de activación, inactivación y recuperación.
Dicha información retroalimenta los modelos moleculares 3D a partir de los cuales se formulan nuevas hipótesis cualitativas sobre el comportamiento de los átomos del canal, causa última subyacente a los fenómenos macroscópicos observables.
Los modelos constituyen “hipótesis atómicas tridimensionales” asociadas a información experimental y cuyo comportamiento y evolución depende de aproximaciones estadísticas de las leyes fisicoquímicas de la naturaleza en un ambiente virtual en el cual se puede explorar una infinidad de posibilidades.
Por lo tanto, estos modelos a su vez contribuyen a dar una explicación coherente a los resultados empíricos y guían el diseño de nuevos experimentos que puedan contestar preguntas de relevancia fisiológica e importancia médica. Incluso bajo el espíritu newtoniano de un universo mecánico, resultaría terriblemente reduccionista e ingenuo dar a esta etérea imagen de canales iónicos, por elaborada y metódica que haya sido su creación, la calidad de causa última de un fenómeno biológico.
Pero bien es cierto que la delicada y extraordinaria comunicación celular que garantiza la vida de toda especie sobre la Tierra depende de un lenguaje que, en esencia, es una oscilación coordinada de cargas eléctricas, lejana al equilibrio termodinámico y fuertemente determinada por la estructura atómica de los canales por los cuales fluyen.
Este es, al menos, un mejor modelo sobre la manera en la cual las células, las unidades fundamentales de la vida, transmiten información e interactúan coordinadamente entre sí para conformar el conjunto de ultraestructuras funcionales que constituyen los organismos. Un modelo falible y limitado al fin.
Sin embargo, en palabras del físico americano Henry A. Bent: “Un modelo debe ser erróneo hasta cierto grado, si no sería la cosa misma que representa. El truco está en ver en qué es correcto”.[11]
REFERENCIAS
1 L'Univers, disait-t-il un jour, est un ensemble dissymétrique. Je suis porté à croire que la vie, telle qu'elle se manifeste à nous, doit être fonction de la dissymétrie de l'Univers ou des conséquences qu'elle entraîne. Works 1, Comptes Rendus de l'Académie des Sciences (1874).
2 Goldbetter A. Biological Rythms as Temporal Dissipative Structures. Special Volume in Memory of Ilya Prigogine. Advances in Chemical Physics, 135. John Wiley & Sons, Inc (2007).
3 Catterall WA. From ionic currents to molecular mechanism: the structure and function of voltage-gated sodium channels. Neuron, 26 (2000) 13-25.
4 Brackenbury WJ and Isom LL. Voltage-gated Na+ channels: Potential for ß subunits as therapeutic targets. Expert Opinion on Therapeutic Targets 12 (2008) 1191-1203.
5 Clare J J. Targeting ion channels for drug discovery. Discovery Medicine 53 (2010).
6 Overington JP, Al-Lazikani B and Hopkins AL. How many drug targets are there? Nat Rev Drug Discov 5 (2006) 993-996.
7 Ekins S, Mestres J and Testa B. In silico pharmacology for drug discovery: applications to targets and beyond. Br J Pharmacol. 152 (2007) 21-37.
8 Takahashi MP and Cannon SC. Enhanced Slow Inactivation by v445m: A Sodium Channel Mutation Associated with Myotonia. Biophysical Journal 76 (1999) 861-868.
9 Lampert A, O'Reilly AO, Reeh P and Leffler A. Sodium channelopathies and pain. Pflugers Arch. 460 (2010) 249-63.
10 Zheng W and Kiss L. Screening Technologies for Ion Channel Targets in Drug Discovery. American Pharmaceutical Review, Ion Channels.
11 Höltje et al. Molecular Modeling: Basic Principles and Applications. Wiley vch, Weinheim, Germany (2003).
angelislas@gmail.com
Eduardo Salinas Stefanon
esalinass@gmail.com
Instituto de Fisiología BUAP Puebla, México.


No hay comentarios:
Publicar un comentario